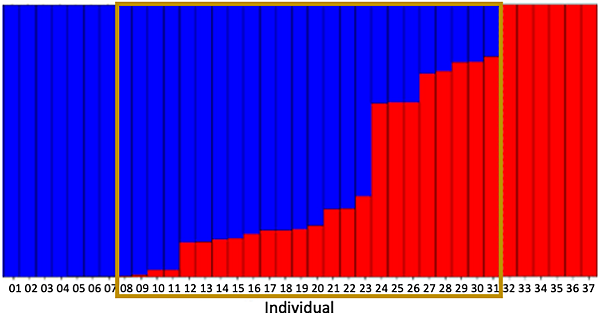
現在生態演化學的大家越來越多使用基因體階級的資料類型了,也越來越多人用這些大量的資料來研究族群分化及物種分化層級的問題。這些都是非常棒的事及發展方向!
族群基因體學研究有一個非常常被使用的軟體Structure(或是ADMIXTURE),它可以根據你輸入的遺傳資料幫你計算出最可能能夠解釋你收集到的資料中的遺傳變異需要有幾個不同遺傳來源的族群(k)。然後再幫你計算每一個你輸入的個體(individual)的遺傳資料有多少百分比是來自各個不一樣的遺傳來源。
airbugs 發表在 痞客邦 留言(0) 人氣(83)
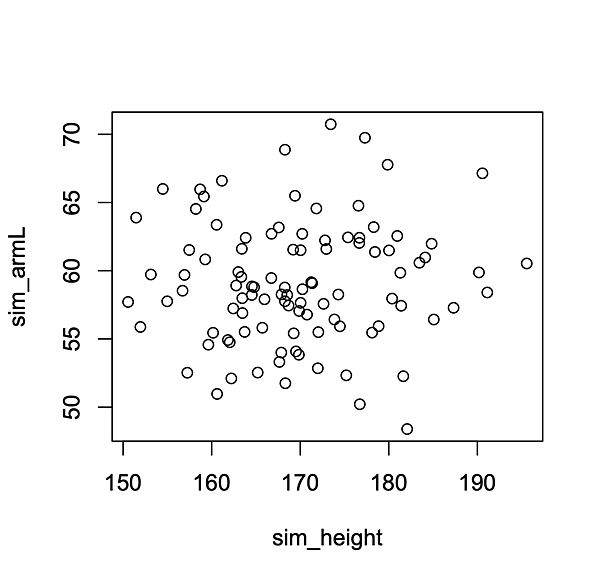
好久沒更新了,來寫一個大家好像不太在意的小地方Spurious Corrlation:
有時候在做生態學研究的時候,因為每個生物個體會有很多本身的差異,所以在做分組比較的時候很多測量值會要標準化。比如鍬形蟲的大顎長度會與體長有關,越大隻的雄蟲越長。鍬形蟲的翅膀長度也會與體長有關,越大隻翅鞘就越長。那如果研究大顎長度與翅鞘長度是不是有相關,能不能把兩個測量值(大顎長度及翅鞘長度)對體長做標準化(變成ratio)在來看兩個比例的關係呢?
airbugs 發表在 痞客邦 留言(0) 人氣(22)
因為我做的研究與物種分化及如何客觀的判別什麼樣的東西可以被稱為不同“種”,不同種與不同“族群”的生物學差異在哪,很多時候都會遇到有興趣的朋友或是同學問說為什麼不試試看雜交?或者是看到不同物種被定義的時候(例如我研究的不同長戟大兜蟲的“種”),常常能得到的反應就是『可是這些不同長戟可以交配生下沒有問題的下一代,為什麼是不同種呢?』。
但是生物種與雜交不孕(或下一代生育率下降)有直接的關係嗎?為什麼很多人(包含生物老師,生物學家及大眾)想到不同生物的種類總是會聯想到雜交不孕呢?
airbugs 發表在 痞客邦 留言(0) 人氣(1,758)

最近常常逛我們博物館的館藏標本,
突然發現Field Museum有好多大王花金龜的標本!!!
最近抽空照了相,但是只挑有"TYPUS"的箱子拍照,
以後也許會有人再重新整理Goliathus的分類學,
airbugs 發表在 痞客邦 留言(1) 人氣(226)
當兩種親緣關係很近的物種分佈在同一個地方的時候,
這兩種物種是如何得以共存呢?
如果他們的食性都差不多,居住偏好及繁殖習慣都一樣,
這兩個物種要不是彼此競爭排除(有一種會絕種),
airbugs 發表在 痞客邦 留言(0) 人氣(474)

距離我前老闆發表Statistical phylogeography這一篇文章已經經過15年了!親緣地理學的研究也慢慢的從之前根據gene tree來講故事變成用電腦模擬來檢測不一樣的歷史模型對所得到分子證據的解釋度。但是既使現在還是可以看到很多文章就是sequence一些loci,重建幾棵gene tree,然後就根據這些基因樹來推測故事...
這樣的方法用在分化很久的不同種之間的親緣關係分析基本上不會有太大的問題,因為基本上lineage都已經sorting的差不多,不同的lineages也已經有累積自己獨特的變異。
但是用於種內的不同個體,或是探討近緣種姊妹種或複合種群內不同個體的關係時,這些gene trees與不同個體之間的geneological history是沒有一定的相關性的。在近期分化的個體之間如果看到不一樣的基因型基本上都是由祖先多態性(ancestral polymorphism)留下來的結果,所以所重建的基因樹歷史並不會與個體間或族群間的分化事件相關(除非你是研究病毒)。用這樣的資料來討論分化的歷史,只是亂看錯誤的圖然後自己編故事。
airbugs 發表在 痞客邦 留言(0) 人氣(510)
Von guten Mächten
--by Dietrich Bonhoeffer
airbugs 發表在 痞客邦 留言(0) 人氣(31)
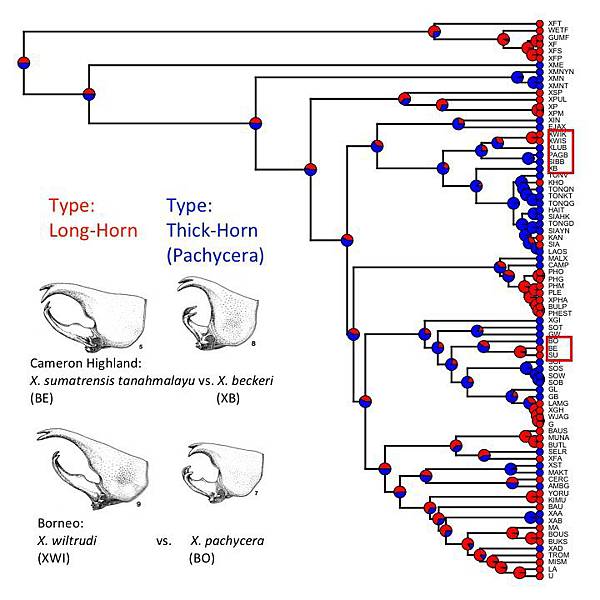
不同類群生物之間的遺傳交流發生的頻率及發生的前提是什麼?
這一直都是演化學研究上未有定論的議題。比如說(一)雜交可能會導致物種的特性消失,最後導致兩個不一樣的生物種混成一種:因此不同生物種間雜交應該是很少發生的事件。但是(二)不同種之間雜交可以讓具有適應性的等位基因(allele)在種間相互交換,可能促進適應性等位基因接受者的類群(allele-receivor lineage)有辦法發育新的表型(phenotype)或進入並使用原本無法使用的生態棲位(niche):因此種間雜交可能會有適應性演化上的好處。論述(二)很好的一個例子就是龍鱗展鬥:人類用雜交的方法把龍鱗這個特徵從另外一個Betta的種轉到splendens這個種裡面,導致他有極高的適存性(fitness),因為人類會繁殖很多有這個特徵的魚體的子代。
但是討論這些問題之前,其實我們並不了解生物種間雜交發生的前提是什麼,或是有哪些因子可能會限制種間雜交發生。今年我用長戟大兜蟲這個類群來專門探討這一類型問題:(一)地理分佈與種間遺傳滲入的關聯性,(二)親緣關係遠近與遺傳滲入發生的關聯性,及(三)型態適應與遺傳滲入的關聯性。
airbugs 發表在 痞客邦 留言(0) 人氣(507)
Mysterium Fidei.(信德的奧跡)
Mortem tuam annuntiamus, Domine,
(基督,我們傳報祢的聖死[祢的死被宣告,我主])
et tuam resurrectionem confitemur,
(我們歌頌祢的復活[及祢的復活被承認])
airbugs 發表在 痞客邦 留言(0) 人氣(58)
如何客觀量化地辨識生物種是生物學上最基本的問題。因為生物學上絕大部分的問題都必須仰賴清楚的分類結果(例如保育政策制定,生態學及演化學的研究都會因為分類學的結果不一樣造成不同的結果)。但是因為生物種的定義百百種,所以既使近年來量化辨識生物種的統計方法發展很多(絕大多數都以分子分類為基礎),拿到同樣的採樣甚至是完全一樣的資料還是可能的到不一樣的的生物種辨識結果
airbugs 發表在 痞客邦 留言(0) 人氣(1,927)





