距離我前老闆發表Statistical phylogeography這一篇文章已經經過15年了!親緣地理學的研究也慢慢的從之前根據gene tree來講故事變成用電腦模擬來檢測不一樣的歷史模型對所得到分子證據的解釋度。但是既使現在還是可以看到很多文章就是sequence一些loci,重建幾棵gene tree,然後就根據這些基因樹來推測故事...
這樣的方法用在分化很久的不同種之間的親緣關係分析基本上不會有太大的問題,因為基本上lineage都已經sorting的差不多,不同的lineages也已經有累積自己獨特的變異。
但是用於種內的不同個體,或是探討近緣種姊妹種或複合種群內不同個體的關係時,這些gene trees與不同個體之間的geneological history是沒有一定的相關性的。在近期分化的個體之間如果看到不一樣的基因型基本上都是由祖先多態性(ancestral polymorphism)留下來的結果,所以所重建的基因樹歷史並不會與個體間或族群間的分化事件相關(除非你是研究病毒)。用這樣的資料來討論分化的歷史,只是亂看錯誤的圖然後自己編故事。
現在可以做族群分化+親緣地理model test的電腦程式越來越多了,有些也越來越平易近人。DIYABC就是其中一個很容易上手的程式。他幾乎所有的分析都可以用“按”的!這讓沒有programing 經驗的研究員方便很多。以下我就用一個簡單的例子來玩玩怎麼檢測不同歷史模型對所得到真實分子資料的解釋度--哪個model對data有最好的fit?
在姬兜蟲(genus Xylotrupes)裡面有一個種叫做siamensis。這一個種在過去被分成很多不一樣的種或是亞種(例如socrates,tonkinensis等等),其最主要的原因就是雄性成蟲的頭角和胸角在不同地理族群之間有很不一樣的發育性狀。例如在泰國大型雄蟲會長很長的胸角並且頭角有個突起(siamensis form),在越南及中國南方和中國西南等地的大型雄蟲則沒有這些性狀,但是通常會長很粗的胸角,頭角前方會往回勾(翹角姬兜tonkinensis form)。這些姬兜基本上都是同一種,生殖器完全相同,因此在Rowland 2011年的名錄裡面整個中國西南(不包含西藏)及中南半島(不包含馬來半島)就只有一種姬兜X. siamensis。
那這時候就有一個很有趣的問題了!這兩群長得很不一樣的X. siamensis到底是怎麼演變出來的呢?

根據生態棲位模擬的結果,現今中國西南,印度阿薩姆及中南半島都是X. siamensis適合的棲地:

但是在上一次大冰河期,X. siamensis只有越南北部及泰國西南部保有兩個主要的合適棲地:
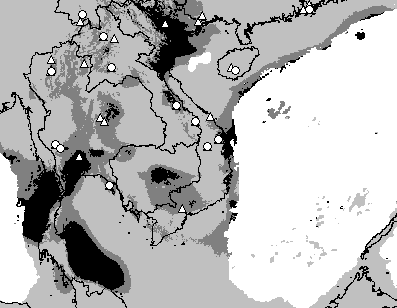
那根據過去的地理分佈變化能不能解釋我們現在看到兩群型態很不一樣的地理族群呢?
現在我手上剛好有三個基因的資料,我把不一樣採樣點的個體歸納到四個地理範圍之中:
Pop1:中國東南及越南,Pop2:雲南(型態像翹角姬兜),Pop3:緬甸靠泰國面的山區(長得像泰國姬兜),Pop4:泰國。
我可以大略得馬上想到三個不一樣的歷史模式來解釋現在看到的兩個地理群:
(一)頭胸角有長有短的是祖先型(來自泰國南部的冰河期避難所),冰河期結束後往東北擴散,然後新族群失去長角特徵。
(二)短角無頭角突的是祖先型(越南北部避難所),冰期結束後往西南擴散,新族群演化出長角特徵!
(三)一個冰河期前廣布的祖先被迫在冰期撤退進入兩個避難所,不同避難所族群分化出不同角特徵,冰期過後再一同擴散進入中國西南及緬甸山區。

下一步就是用DIYABC來分析以上三個不一樣的模型哪一個對我的分子資料有最好的解釋度!
用DIYABC開一個新的MSS的資料夾,之後所有結果都會存在裡面:

然後就是導入sequence alignment的資料。這大概是使用這個程式最麻煩的地方,他有自己的format:
第一行是說雌雄比例為1:1
然後有三個基因 h3,its2,及co1。
h3及its2為核基因 A 代表autosomal
co1為粒線體 M 代表mitochondrial
之後用Pop來區分不同族群的個體,個體名稱與sequences之間用 , 分開。
不同基因需放在<>裡面相互隔開。同基因的不同alleles放在[]裡面相互隔開。
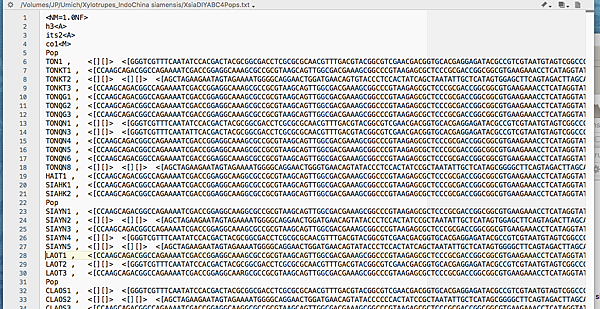
如果你的format都正確,倒入mss file之後會進入下面這一個畫面:
我通常先開始設定歷史模型,把historical model下面的set按下去就好。

右邊的add scenario按鈕可以讓您輸入增加更多的歷史模型,但是在這裡我玩三個就好。
這裡的Scenario 1, 2, 3剛好對應我上面想到的三個歷史模型(如果想知道N1, N2 varNe等argument的意思請參照DIYABC的手冊):

如果你不確定自己arguments寫得對不對,可以按右邊的check and draw scenario鍵來看看:
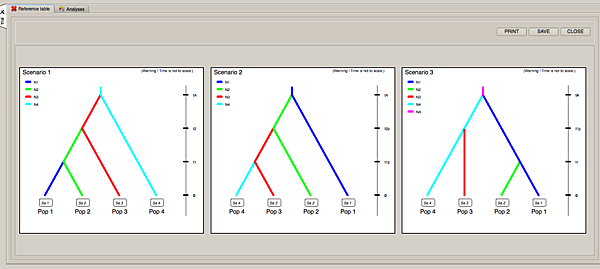
在確定了這三個不一樣的分化過程就是對應我的三個不同歷史模型之後,下一步就是設定priors:
這一步很重要,如果用不對的族群量大小或分化時間的範圍來做電腦模擬,那結果就不可信,切記!
prior setting是ABC分析裡面最重要的一環,設錯了就全錯了!!!

ABC的分析絕對沒有跑一次就可以了。我的例子用default setting的effective pop prior,但是初步跑完後都需要再改:
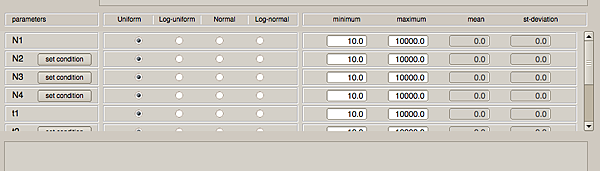
Historical model跟priors設定好了之後就要換到設定Genetic data的選項:
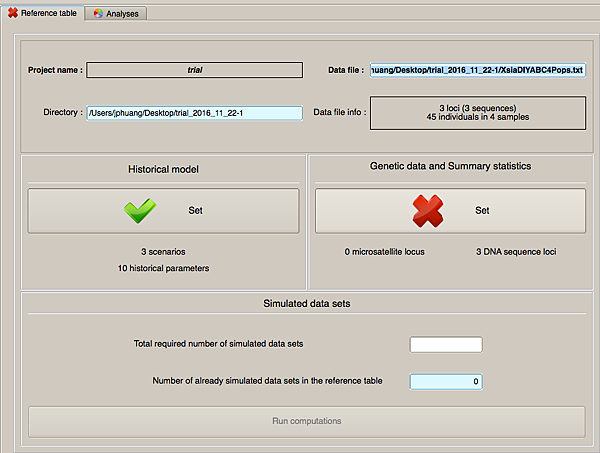
我首先把我的分子資料分成兩個群,一群核基因(h3 + its2)跟另一群粒線體基因(co1):
之後需要分別設這兩個群的分子演化模型(modeltest的結果),即決定要選用哪些population genetics的summary statistics來協助MCMC search(檢測電腦模擬的結果是不是好)。

以粒線體co1這個群組為例,我設定了TN+I+G的演化模型,然後summary statistics我選了tajima's D跟各族群間兩兩比較的FST:


當然你可以選很多很多的summary satistics來假裝你的電腦simulation一定要很接近的結果你才要。但是要記住這樣的話你的電腦模擬可以永遠都得不到任何接近真實data的結果,或是你要simulate非常非常久才能得到一點點可以用的資料。決定權在個人,你有辦法說服reviewers就好。
該設定的東西都設定好之後就按下validation 鍵,沒問題就會回到主畫面:

下一步就是決定MCMC要跑多久,我的例子就照這個program給我的建議好3百萬代(每個historical model各1百萬代MCMC):
program人很好的跟我說大概要跑14分鐘。
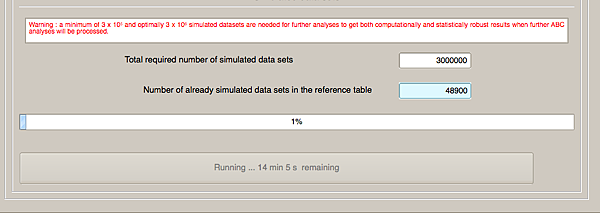
跑完MCMC模擬三百萬代之後終於可以分析資料啦~終於可以按下Analyses這一個tab:

首先根據我們真實得到的資料跟電腦模擬資料計算出來的summary statistics跑個PCA:

我的結果還好,模擬資料的結果(小點點)都還在真實資料(黃色大點點)的附近,不會差太遠:

接下來就是做model comparison!!!我們最想知道的結果:
根據一定的誤差範圍(自己定可以接受的範圍),歷史模型(二)最常產生接近真實資料的結果!
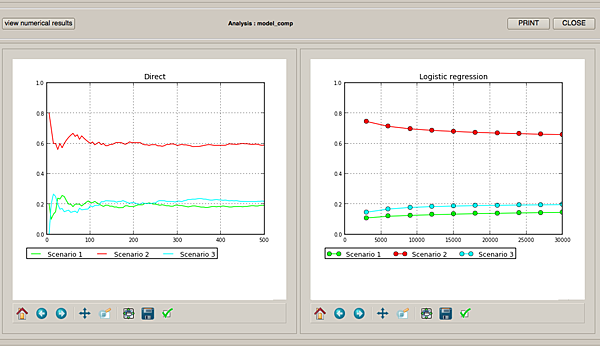
到這裡,很高興了!得到了結果顯示(二)是三個歷史模型中最可能的model!!
但是這樣就結束了嗎?就可以說小角角會生出大角角的後代的嗎?
當然不是...............
來看看族群參數的估計結果吧(紅色是prior綠色是posterior,只用model 2的模擬結果):
有效族群量:

朔源時間:
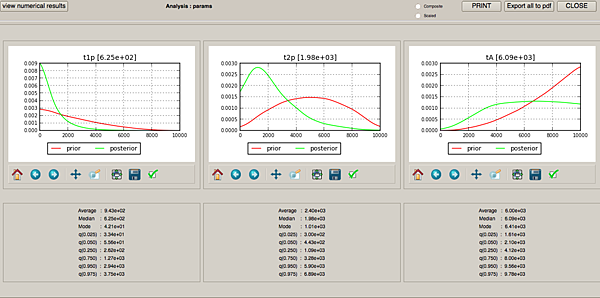
有效族群量N1 N2等等的posterior都爛斃了...prior設太小了....
想要發表的話就在重跑一次吧,但是uniform prior的上限要開大一點。
其實可以先用 IM或Migrate-N等等program先估計好這些族群遺傳的參數(就是Ne跟tau),然後再根據這些估計結果設定prior,會比較快,不用像我這個例子一樣亂亂試。
就醬,abc的分析已經不難做了!大家對親緣地理,族群分化,種化等等議題有興趣的話,試試ABC吧!
這樣的方法用在分化很久的不同種之間的親緣關係分析基本上不會有太大的問題,因為基本上lineage都已經sorting的差不多,不同的lineages也已經有累積自己獨特的變異。
但是用於種內的不同個體,或是探討近緣種姊妹種或複合種群內不同個體的關係時,這些gene trees與不同個體之間的geneological history是沒有一定的相關性的。在近期分化的個體之間如果看到不一樣的基因型基本上都是由祖先多態性(ancestral polymorphism)留下來的結果,所以所重建的基因樹歷史並不會與個體間或族群間的分化事件相關(除非你是研究病毒)。用這樣的資料來討論分化的歷史,只是亂看錯誤的圖然後自己編故事。
現在可以做族群分化+親緣地理model test的電腦程式越來越多了,有些也越來越平易近人。DIYABC就是其中一個很容易上手的程式。他幾乎所有的分析都可以用“按”的!這讓沒有programing 經驗的研究員方便很多。以下我就用一個簡單的例子來玩玩怎麼檢測不同歷史模型對所得到真實分子資料的解釋度--哪個model對data有最好的fit?
在姬兜蟲(genus Xylotrupes)裡面有一個種叫做siamensis。這一個種在過去被分成很多不一樣的種或是亞種(例如socrates,tonkinensis等等),其最主要的原因就是雄性成蟲的頭角和胸角在不同地理族群之間有很不一樣的發育性狀。例如在泰國大型雄蟲會長很長的胸角並且頭角有個突起(siamensis form),在越南及中國南方和中國西南等地的大型雄蟲則沒有這些性狀,但是通常會長很粗的胸角,頭角前方會往回勾(翹角姬兜tonkinensis form)。這些姬兜基本上都是同一種,生殖器完全相同,因此在Rowland 2011年的名錄裡面整個中國西南(不包含西藏)及中南半島(不包含馬來半島)就只有一種姬兜X. siamensis。
那這時候就有一個很有趣的問題了!這兩群長得很不一樣的X. siamensis到底是怎麼演變出來的呢?

根據生態棲位模擬的結果,現今中國西南,印度阿薩姆及中南半島都是X. siamensis適合的棲地:

但是在上一次大冰河期,X. siamensis只有越南北部及泰國西南部保有兩個主要的合適棲地:

那根據過去的地理分佈變化能不能解釋我們現在看到兩群型態很不一樣的地理族群呢?
現在我手上剛好有三個基因的資料,我把不一樣採樣點的個體歸納到四個地理範圍之中:
Pop1:中國東南及越南,Pop2:雲南(型態像翹角姬兜),Pop3:緬甸靠泰國面的山區(長得像泰國姬兜),Pop4:泰國。
我可以大略得馬上想到三個不一樣的歷史模式來解釋現在看到的兩個地理群:
(一)頭胸角有長有短的是祖先型(來自泰國南部的冰河期避難所),冰河期結束後往東北擴散,然後新族群失去長角特徵。
(二)短角無頭角突的是祖先型(越南北部避難所),冰期結束後往西南擴散,新族群演化出長角特徵!
(三)一個冰河期前廣布的祖先被迫在冰期撤退進入兩個避難所,不同避難所族群分化出不同角特徵,冰期過後再一同擴散進入中國西南及緬甸山區。

下一步就是用DIYABC來分析以上三個不一樣的模型哪一個對我的分子資料有最好的解釋度!
用DIYABC開一個新的MSS的資料夾,之後所有結果都會存在裡面:

然後就是導入sequence alignment的資料。這大概是使用這個程式最麻煩的地方,他有自己的format:
第一行是說雌雄比例為1:1
然後有三個基因 h3,its2,及co1。
h3及its2為核基因 A 代表autosomal
co1為粒線體 M 代表mitochondrial
之後用Pop來區分不同族群的個體,個體名稱與sequences之間用 , 分開。
不同基因需放在<>裡面相互隔開。同基因的不同alleles放在[]裡面相互隔開。

如果你的format都正確,倒入mss file之後會進入下面這一個畫面:
我通常先開始設定歷史模型,把historical model下面的set按下去就好。

右邊的add scenario按鈕可以讓您輸入增加更多的歷史模型,但是在這裡我玩三個就好。
這裡的Scenario 1, 2, 3剛好對應我上面想到的三個歷史模型(如果想知道N1, N2 varNe等argument的意思請參照DIYABC的手冊):

如果你不確定自己arguments寫得對不對,可以按右邊的check and draw scenario鍵來看看:

在確定了這三個不一樣的分化過程就是對應我的三個不同歷史模型之後,下一步就是設定priors:
這一步很重要,如果用不對的族群量大小或分化時間的範圍來做電腦模擬,那結果就不可信,切記!
prior setting是ABC分析裡面最重要的一環,設錯了就全錯了!!!

ABC的分析絕對沒有跑一次就可以了。我的例子用default setting的effective pop prior,但是初步跑完後都需要再改:

Historical model跟priors設定好了之後就要換到設定Genetic data的選項:

我首先把我的分子資料分成兩個群,一群核基因(h3 + its2)跟另一群粒線體基因(co1):
之後需要分別設這兩個群的分子演化模型(modeltest的結果),即決定要選用哪些population genetics的summary statistics來協助MCMC search(檢測電腦模擬的結果是不是好)。

以粒線體co1這個群組為例,我設定了TN+I+G的演化模型,然後summary statistics我選了tajima's D跟各族群間兩兩比較的FST:


當然你可以選很多很多的summary satistics來假裝你的電腦simulation一定要很接近的結果你才要。但是要記住這樣的話你的電腦模擬可以永遠都得不到任何接近真實data的結果,或是你要simulate非常非常久才能得到一點點可以用的資料。決定權在個人,你有辦法說服reviewers就好。
該設定的東西都設定好之後就按下validation 鍵,沒問題就會回到主畫面:

下一步就是決定MCMC要跑多久,我的例子就照這個program給我的建議好3百萬代(每個historical model各1百萬代MCMC):
program人很好的跟我說大概要跑14分鐘。

跑完MCMC模擬三百萬代之後終於可以分析資料啦~終於可以按下Analyses這一個tab:

首先根據我們真實得到的資料跟電腦模擬資料計算出來的summary statistics跑個PCA:

我的結果還好,模擬資料的結果(小點點)都還在真實資料(黃色大點點)的附近,不會差太遠:

接下來就是做model comparison!!!我們最想知道的結果:
根據一定的誤差範圍(自己定可以接受的範圍),歷史模型(二)最常產生接近真實資料的結果!

到這裡,很高興了!得到了結果顯示(二)是三個歷史模型中最可能的model!!
但是這樣就結束了嗎?就可以說小角角會生出大角角的後代的嗎?
當然不是...............
來看看族群參數的估計結果吧(紅色是prior綠色是posterior,只用model 2的模擬結果):
有效族群量:

朔源時間:

有效族群量N1 N2等等的posterior都爛斃了...prior設太小了....
想要發表的話就在重跑一次吧,但是uniform prior的上限要開大一點。
其實可以先用 IM或Migrate-N等等program先估計好這些族群遺傳的參數(就是Ne跟tau),然後再根據這些估計結果設定prior,會比較快,不用像我這個例子一樣亂亂試。
就醬,abc的分析已經不難做了!大家對親緣地理,族群分化,種化等等議題有興趣的話,試試ABC吧!
全站熱搜


 留言列表
留言列表
